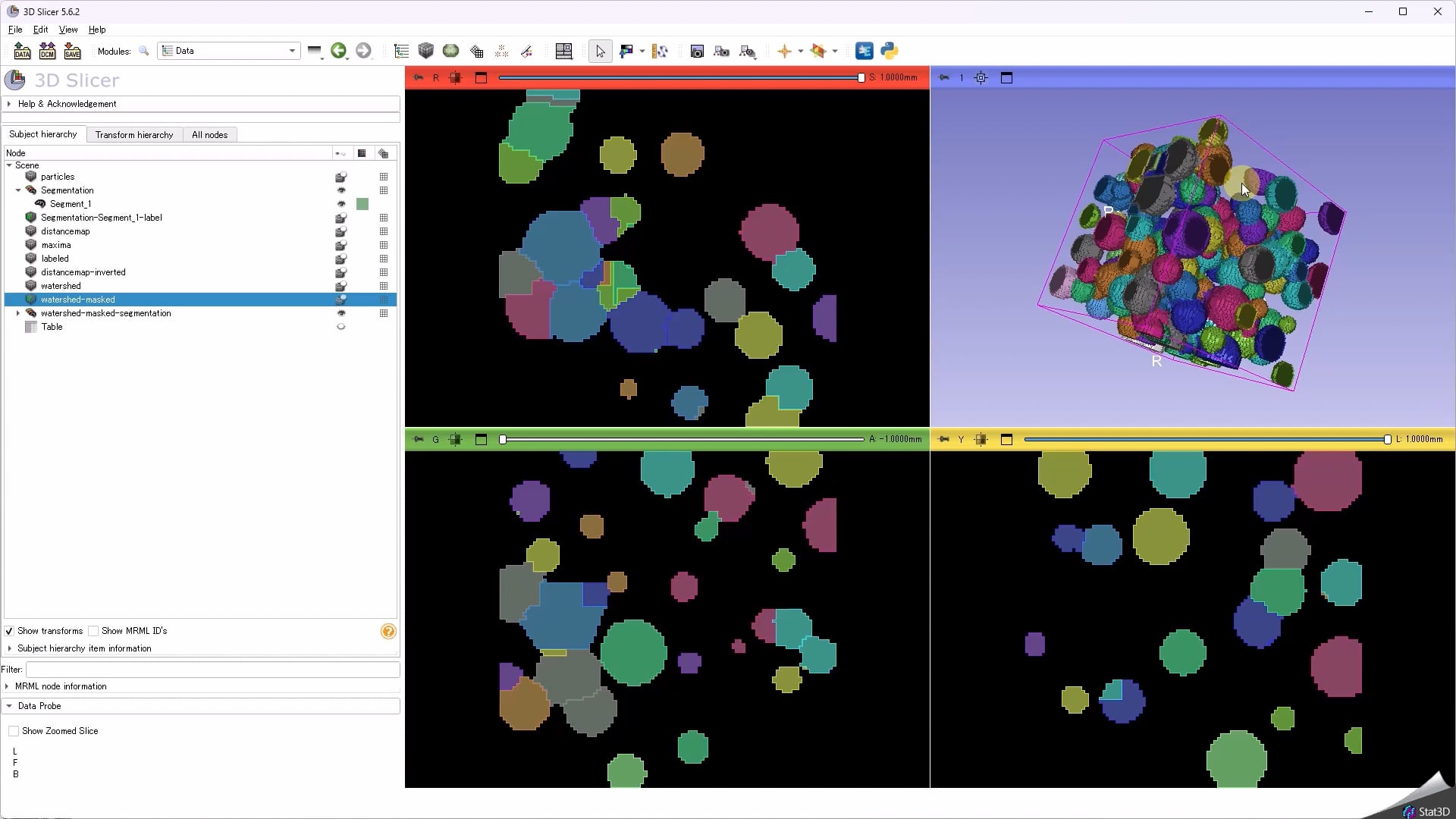
動画の概要
この動画では、X線CT等で撮影した粒子状のデータを想定して、フリーの3次元画像処理ソフトウェアである3D Slicerを用いて、くっついている粒子どうしを分割する方法を説明しています。分割には距離変換と極大点抽出、Watershedといった手法を用います。また、分割した後の各粒子の体積を計算する方法も説明しています。
ポイント
- Segmentation Editorでグレースケール画像を元にセグメンテーション
- Thresholdツール
- Applyで確定
- ラベルデータに変換
- Dataモジュールで、「Export visible segment to binary label map」をクリック
- Simple Filtersモジュールで変換していく
- Simple Filtersでは、ラベルとグレースケールの区別をしないので注意
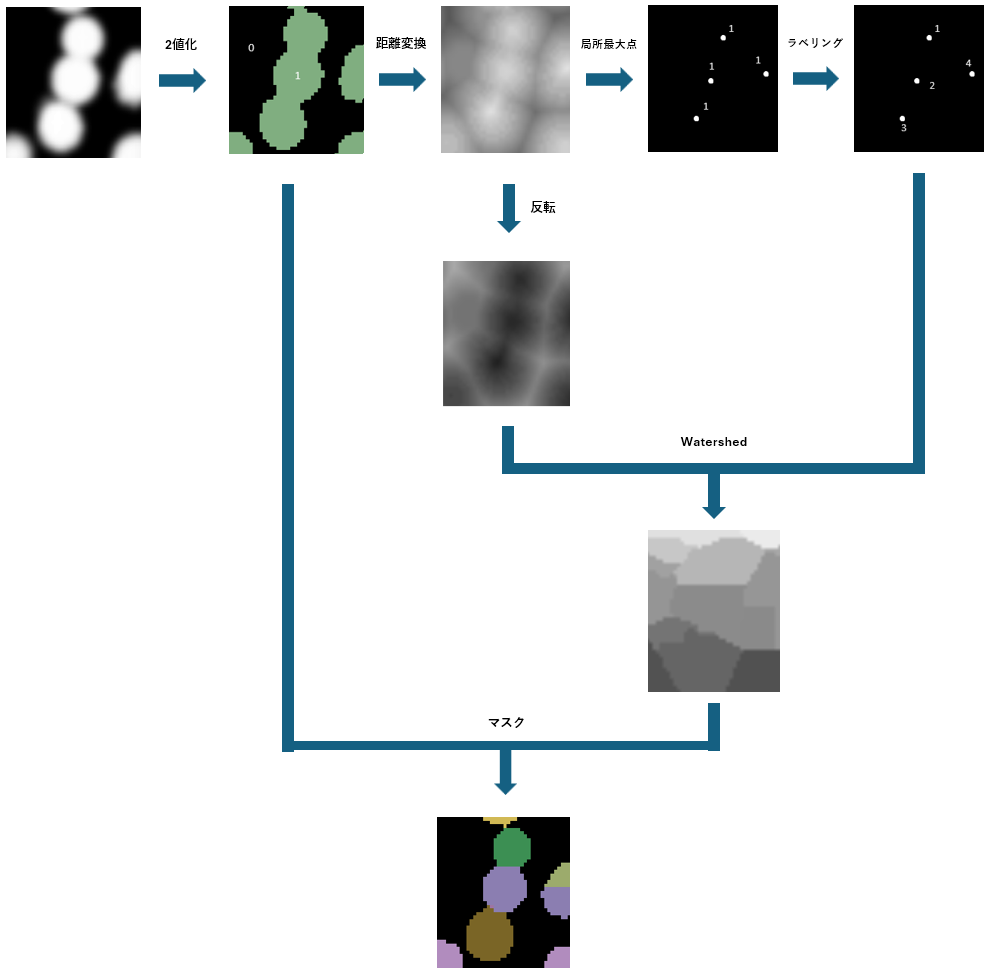
- マーカー画像の生成
- SignedDanielssonDistanceMapImageFilter: 距離マップを生成
- Inside Is Positiveにチェックを入れる
- RegionalMaximaImageFilter: 局所最大値を取り出す
- Fully Connectedはチェックを入れたほうがノイズ抑制できる
- BinaryDilateImageFilter: (必要な場合のみ)近い位置にある局所最大値を接続することで、分割の細かさを下げることができる
- ConnectedComponentImageFilter: 局所最小値ごとに別々の値を付与
- SignedDanielssonDistanceMapImageFilter: 距離マップを生成
- 勾配画像の生成
- InvertIntensityImageFilter: Watershedの入力として使うために、距離画像の反転画像を生成(粒子の境界がはっきりしている場合は、GradientMagunitudeImageFilterで元画像の勾配を計算して使ってもよい)
- Watershedの実行
- MorphologicalWatershedFromMarkersImageFilter: マーカーを起点に領域を拡大
- 元のラベル画像でマスク
- MaskImageFilter: ラベルの外側を0で埋める
- グレースケールからラベルに変換
- VolumesモジュールでVolume Informationを開き、Convert to label mapでConvertを実行
- セグメンテーションデータに変換
- Dataモジュールで、「Convert labelmap to segmentation node」をクリック
- Segment Statisticsで体積などを計算
- テーブルが表示される